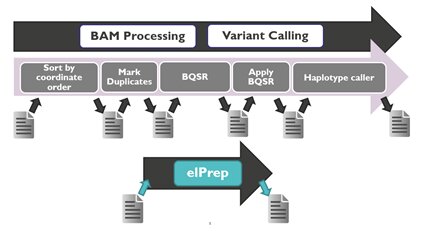
图1:变异检测的流程一般由几个连续的步骤所组成,而每一步都需要占用计算机内存进行读写。本文所开发的新软件elPrep将变异检测流程中的所有步骤合并成一步,通过并行化处理加快运算。(图片来源:比利时微电子研究中心)
尽管当今(基因)测序成本已极大程度地降低了,但它依旧无法被纳入常规分析中。其中一个原因是原始测序数据的处理需要耗费大量时间和大量昂贵的计算资源,如:全基因测序数据的分析需要几天时间完成,数据分析所需的计算平台需租赁数据中心昂贵的服务器。
比利时微电子研究中心ExaScience Life实验室的Charlotte Herzeel指出:“现有的测序数据分析和变异检测软件并非以最优化的方式构建框架。在变异检测流程中,每一步都需单独调用其他软件工具执行,这会额外消耗内存进行读写工作,且仅当上一步任务完成后,下一步才可进行。而我们开发的新软件,重新考虑了整个分析流程,将所有步骤作为一个整体。”
elPrep5:通过并行化高效进行变异检测
elPrep5是比利时微电子研究中心ExaScience Life实验室为测序数据分析开发的最终版软件。这次版本更新的内容包括了变异检测功能,而这一步通常会占用整个分析流程中38-80%的运行时间。elPrep是基于Linux系统开发、并用Go语言编写的软件(Go语言是Google公司开发的一种新计算机语言)。该版本已在Github平台上公开发布,同时也已获得了高级许可证的支持。
Herzeel解释道:“本软件的优化策略在于将多步操作合并成一步,进行并行化处理分析,同时避免生成I/O文件(读写文本文件)。该软件运行的结果可与当前最先进的基因组分析软件媲美,如SAMtools,Picard和GATK4。因此,用户可将elPrep5当作其他热门工具的临时替代软件。”
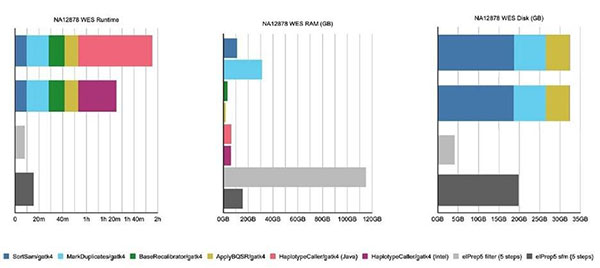
图2. GATK4和elPrep软件分别对全外显子组数据评估测试实验的对比结果。(图片来源:比利时微电子研究中心)
加速分析:分析速度提高至8-16倍
为了测试elPrep5软件的性能,研究人员进行了很多评估实验。Herzeel提及道:“第一个实验使用了96CPUx384GB配置的服务器进行了全外显子组序列的分析。这些数据要么使用常规的GATK4软件进行分析,要么使用elPrep软件。我们使用了GATK4软件的两种模式进行分析:一种是Java模式(使用标准的haplotype caller变异位点检测算法),另一种是Intel模式(优化版并行化处理的算法)。对于elPrep,我们也对两种分析模式进行了测试:一种是filter模式(将所有文件读取于内存中,从而避免在磁盘中产生I/O中间文件),另一种是sfm(将数据集按染色体位置分区)。总的来说,elPrep软件的filter模式因消耗大量内存,故更适用于小数据集的分析。因此能否使用filter模式进行分析往往取决于输入文件BAM的大小以及可用内存量的大小。”
此实验的结果展示在图2和图3中。
elPrep软件的filter模式下对全外显子组数据进行分析得到了以下结果:运行速度比GATK4(Java和Intel模式)快11-15倍;内存使用量比GATK4(Java和Intel模式)多4倍;磁盘空间消耗量约是GATK4 (Java和Intel模式)的十分之一。
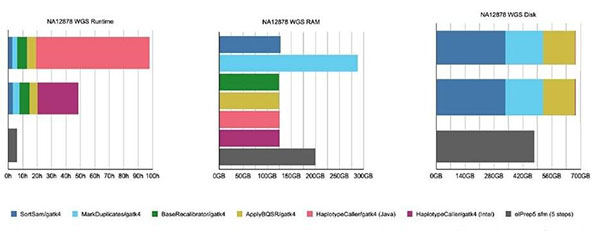
图3. GATK4和ePrep软件分别对全基因组数据评估测试实验的对比结果。(图片来源:比利时微电子研究中心)
elPrep软件的sfm模式下对外显子组数据进行分析得到了以下结果:运行速度比GATK4(Java和Intel模式)快6到7.5倍;内存使用量比GATK4少一半(Java和Intel模式);磁盘空间消耗量比GATK少一半(Java和Intel模式)。
Herzeel解释道:“基于云端的条件下,elPrep软件的filter模式总体来说是性价比最高的。尽管它的内存使用量比sfm模式要多,但运行时间却大大降低,相对而言减少了服务器的租赁成本。”类似的对比实验也在全基因组数据上进行了,唯一的不同点在于elPrep软件只能用sfm模式进行分析,filter模式并不适用于庞大的全基因组数据集。
elPrep软件的sfm模式下对基因组数据进行分析得到了以下结果:运行速度比GATK4的Java模式快16倍,比GATK4的Intel模式快8.5倍;内存使用量比GATK4(Java和Intel模式)少30%;磁盘空间消耗量比GATK4节省30%(Java和Intel模式)。
Herzeel总结道:“elPrep5对全基因组数据的分析速度比全外显子组数据快16倍。这是因为elPrep5软件包含了变异位点检测的功能,而正是变异位点的检测通常在全基因组数据的分析中占用了大部分的运行时间。因此,常规全基因组数据集的分析中变异位点检测的运行时间占比越高,elPrep提升的速度就越多。”
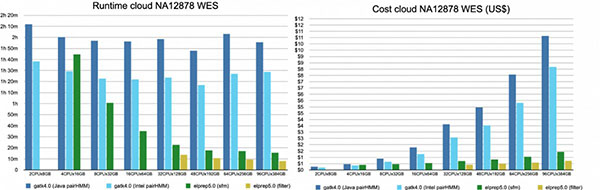
图4. 基于不同型号服务器的条件下,不同软件、不同模型对全外显子组数据分析的运行时间和成本的对比。(图片来源:比利时微电子研究中心)
使用更多的资源意味着实现更快的运行速度(elPrep)
为了测试使用更多的硬件资源时,elPrep可以减少的运行时间,研究人员进一步评估了elPrep的运行潜能 。Herzeel评价道:“假设该软件能实现上述功能,那么减少的运行时间可以弥补租赁高性能服务器的高成本。为了实现此次测试,我们以常用的GATK4软件作为对比,在配置了不同的CPU和内存的服务器上进行了变异位点检测。”
图4、5分别展示的是全外显子组和全基因组的分析结果。
对于全外显子组数据,elPrep软件(filter和sfm模式)运行妥当。每增加一个运算资源,运行时间会相对的减半。从图中可得出具体的结果:
elPrep软件的filter模式在96CPUx384GB配置下运行速度最快,比GATK4软件Intel模式在48CPUx192GB配置下的最快速度还要快上10倍,且价格仅为五分之一。
基于2CPUx8GB配置下运行GATK4软件Intel模式的运算是最划算的。然而,此规划却比elPrep最高速运行下慢12倍,尽管其价格仅为elPrep的四分之一。相对于最便宜的GATK4软件的运行成本,花两倍的价格即可在基于8CPUx32GB的配置下运行elPrep软件的sfm模式,速度比GATK4快上2倍。
如果用户更倾向于使用GATK4软件Java模式,那么选择使用elPrep软件会更便宜,并且运行速度更快。在32CPUx128GB配置下运行elPrep软件的filter模式会比在2CPUx8GB配置下运行GATK4软件的Java模式相对便宜,且运行速度快将近9倍。
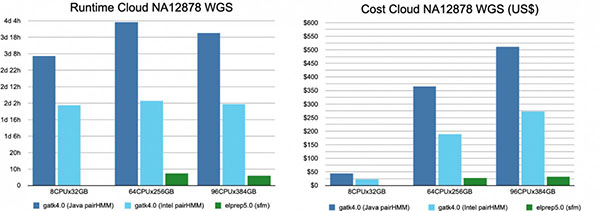
图5. 基于不同型号服务器的条件下,不同软件的不同模型对全基因组数据分析运行时间和成本的对比。(图片来源:比利时微电子研究中心)
对于庞大的全基因组数据而言,elPrep软件只有sfm模式能在足够的内存和磁盘空间配置的服务器上运行。我们从图4可以发现一个有趣的现象:在96CPUx384GB配置下运行elPrep软件比在8CPUx32GB配置下运行GATK4软件Java模式要便宜,这是因为elPrep软件的运行速度比GATK4软件快将近14倍。具体来说,由于运行时间从将近80个小时降低到6小时以下,运行成本从45美元降低到了32美元。
除了使用大型云商提供的服务器之外,我们还在使用不同的存储架构(FlashBlade)的Pure Storage基础设施的系统上进行了测试。测试结果如图6。
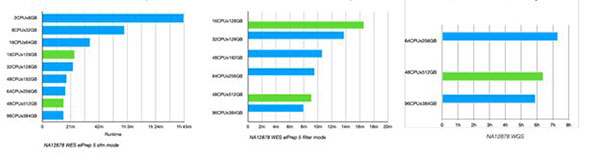
图6. 在云端(蓝色)和Pure Storage(绿色)(使用的Interl和AMD配置的服务器)上运行elPrep的实验结果。服务器根据内存量和CPU核数进行排名。云商一般将数据存储在默认的热数据存储器上,而Pure Storage平台将数据存储在与计算服务器相连的Flashblade服务器上。左边的两幅图显示的是全外显子组数据的分析结果,最右边的图显示的是全基因组数据的分析结果。
Herzeel表示:“elPrep软件在Pure Storage平台上的运行效果与在云端上相当,这表明Pure Storage平台的Flashblade存储方案运行效果至少与云商的存储方案旗鼓相当。”
elPrep软件的最新版本elPrep5是一款用于测序数据分析的软件,它还可以进行变异位点检测。此软件可替代当前最先进的基因组分析程序,如SAMtools, Picard和GATK4。
Herzeel总结道:“我们的评估测试实验表明,与GATK4软件相比,elPrep5软件的运行速度快8到16倍。具体来说,在处理全基因组数据时,elPrep5软件的变异位点检测功能耗时减少至6小时以内,而对全外显子组数据而言,仅需不到8分钟即可完成处理。elPrep软件之所以运行速度如此之快,在于创新的算法和并行化的运算;同时该软件的提速在普通的CPU配置的服务器上也可实现,无需专门的加速器辅助,并且消耗更少的内存和磁盘空间。”